Bernhard
Peter
Aufbau von
Influenza-Viren:
Erkennungsmolekül Hämagglutinin
Hämagglutinin ist ein Glykoprotein, das an 2-3 Stellen mit einem Zuckerrest verknüpft ist. Das Molekulargewicht beträgt ca. 76000. Es ist das hauptsächliche Antigen von Influenza-Viren und so in der Lipidmembran verankert, daß sich der größte Teil des Moleküls mit mindestens 5 verschiedenen antigenen Domains (A, B, C, D, E) in einer Art kugelförmigem Kopf außerhalb des Virus befindet.
Hämagglutinin hat zwei Funktionen:
Das Hämagglutinin ist verantwortlich für die Erkennung der Zielzelle. Seine genaue Aminosäure-Abfolge und seine Raumstruktur ist entscheidend dafür verantwortlich, welche Wirts-Spezifität die betreffende Influenza-Variante hat, ob sie z. B. Vögel infiziert oder Menschen. Die Hämagglutinin-Moleküle sind zu Homo-Trimeren assoziiert und in die Lipiddoppelmembran der Virushülle verankert. Wie Spikes stehen sie aus ihr heraus und verleihen dem Virus das Aussehen eines molekularen Morgensternes.

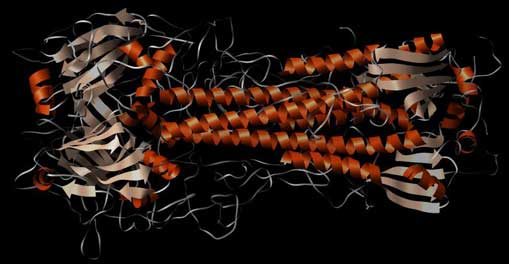
Abb.: Homo-Trimer des Hämagglutinins von Influenza-Viren. Dargestellt ist die Sekundär-Struktur unter Hervorhebung der Alpha-Helices und der Beta-Faltblatt-Strukturen. Man beachte die extrem langen Helices im „Schaft“ des Moleküls. PDB-ID: 4HMG.
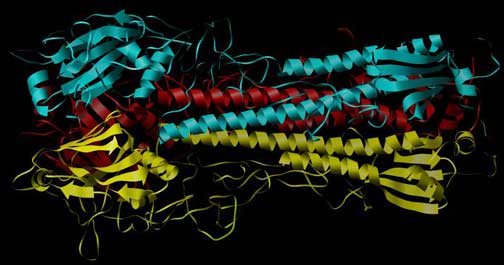
Abb.: Alternative Darstellung des Homo-Trimers von Hämagglutinin von Influenza-Viren. Dargestellt ist die Sekundär-Struktur unter Hervorhebung der Alpha-Helices und der Beta-Faltblatt-Strukturen. Man beachte die Struktur als Assoziat aus drei identischen Untereinheiten (Homo-Trimer), hier in blau, rot und gelb dargestellt). PDB-ID: 4HMG.
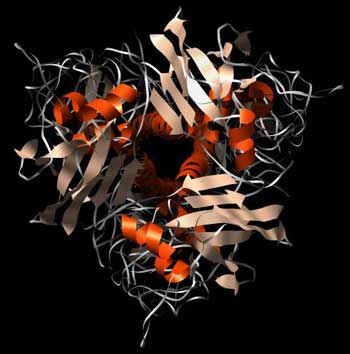
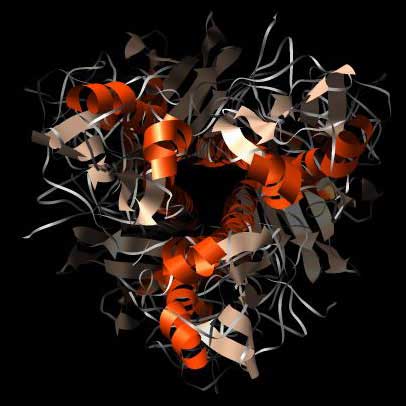
Abb.: Darstellung des Homo-Trimers von Hämagglutinin von Influenza-Viren. Der Blick auf das dickere (links) und dünnere (rechts) Ende des Assoziates illustriert den symmetrischen Aufbau aus drei identischen Untereinheiten (Homo-Trimer). PDB-ID: 3HMG.
Ein niedriger pH-Wert induziert beim Hämagglutinin des Influenza-Virus eine Konformationsänderung, worauf das Hämagglutinin als Mediator für die Fusion der Lipiddoppelmembranen von Virus und Wirtszelle fungiert. Die dreidimensionale Struktur eines Fragmentes von Hämagglutinin zeigt eine größere Umsortierung der Sekundär- und Tertiärstruktur des Moleküles: Das apolare Fusionspeptid bewegt sich dabei um ca. 100 A zur Molekülspitze. Am anderen Ende des Moleküls entfaltet sich ein helikales Segment, eine Sub-Domain ändert komplett ihre Raumposition und klappt so um, daß sich sogar die Richtung der Kette im Raum ändert.
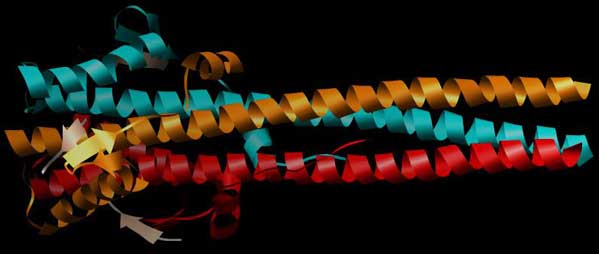
Abb.: Darstellung des Homo-Trimers von Hämagglutinin von Influenza-Viren beim pH-Wert der Membranfusion. Die Abbildung zeigt die konformativen Änderungen, die bei der Vorbereitung der Membranfusion erfolgen. PDB-ID: 1HTM.
Die exakte Raumstruktur von Hämagglutinin ist für die Wirtsspezifität verantwortlich. Mutationen in der Abfolge der Aminosäuren entscheiden darüber, ob eine Virus-Variante auf Vögel beschränkt bleibt oder auf den Menschen überspringt. Es gibt 15 verschiedene Subtypen des Influenza A-Virus in Bezug auf das Hämagglutinin, H1-H15. Alle 15 kommen bei Vögeln vor. Drei davon verursachten im 20. Jh. Pandemien: H1 im Jahre 1918 (und 1977), H2 im Jahre 1957 und H3 im Jahre 1968. 1997 verursachte ein H5-Vogelvirus und 1999 ein H9-Vogelvirus jeweils einen Outbreak in Hongkong. Momentan erhitzt wieder das Vogel-Virus H5N1 die Gemüter. Das verantwortliche Hämagglutinin H5 sieht folgendermaßen aus:

Abb.: Darstellung eines Monomers von H5-Hämagglutinin von Influenza-Viren, wie sie derzeit als Vogelgrippe für Aufregung sorgt. PDB-ID: 1JSM.
Grundlage für obige Abbildungen:
Research Collaboratory for Structural
Bioinformatics Protein Data Bank http://www.rcsb.org/pdb, PDB-ID:
4HMG Weis, W.I., Bruenger, A.T., Skehel, J.J., Wiley, D.C.
REFINEMENT OF THE INFLUENZA VIRUS HEMAGGLUTININ BY SIMULATED
ANNEALING, Originalbeitrag: Weis, W.I., Brunger, A.T., Skehel,
J.J., Wiley, D.C. Refinement of the influenza virus hemagglutinin
by simulated annealing. J. Mol. Biol. ![]() v212
v212 ![]() pp.737-761, 1990
pp.737-761, 1990![]() . Erzeugt mit dem
Programm X-PLOR.
. Erzeugt mit dem
Programm X-PLOR.
Research Collaboratory for Structural
Bioinformatics Protein Data Bank http://www.rcsb.org/pdb, PDB-ID:
3HMG, Weis, W.I., Brunger, A.T., Skehel, J.J., Wiley, D.C:
REFINEMENT OF THE INFLUENZA VIRUS HEMAGGLUTININ BY SIMULATED
ANNEALING, Originalbeitrag: Weis, W.I., Brunger, A.T., Skehel,
J.J., Wiley, D.C. Refinement of the influenza virus hemagglutinin
by simulated annealing. J. Mol. Biol. ![]() v212 pp.737-761,
1990. Erzeugt mit dem Programm X-PLOR. Abbildung erzeugt mit dem
Programm Chimera.
v212 pp.737-761,
1990. Erzeugt mit dem Programm X-PLOR. Abbildung erzeugt mit dem
Programm Chimera.
Research Collaboratory for Structural
Bioinformatics Protein Data Bank http://www.rcsb.org/pdb, PDB-ID:
1HTM, Bullough, P.A., Hughson, F.M., Skehel, J.J., Wiley, D.C.
STRUCTURE OF INFLUENZA HAEMAGGLUTININ AT THE PH OF MEMBRANE
FUSION (Homotrimer), Originalbeitrag: Bullough, P.A., Hughson,
F.M., Skehel, J.J., Wiley, D.C. Structure of influenza
haemagglutinin at the pH of membrane fusion. ![]() Nature
Nature ![]() v371
v371 ![]() pp.37-43, 1994
pp.37-43, 1994
Research Collaboratory for Structural
Bioinformatics Protein Data Bank http://www.rcsb.org/pdb, PDB-ID:
1JSM, Ha, Y., Stevens, D.J., Skehel, J.J., Wiley, D.C. STRUCTURE
OF H5 AVIAN HAEMAGGLUTININ, Originalbeitrag: Ha, Y., Stevens,
D.J., Skehel, J.J., Wiley, D.C. H5 avian and H9 swine influenza
virus haemagglutinin structures: possible origin of influenza
subtypes. ![]() EMBO J.
EMBO J. ![]() v21
v21 ![]() pp.865-875, 2002
pp.865-875, 2002
Aufbau
von Influenza-A-Viren
- Das Enzym der Freiheit: Neuraminidase - Der natürliche Ligand
der Neuraminidase: Sialinsäure
Das M1-Struktur-Protein - Der Mechanismus der
Vermehrung - Launenhafte
Viren: Antigen-Drift und Antigen-Shift - Literatur, Quellen und
Links
Zurück zur Übersicht Grippe /
Influenza
Andere pharmazeutische Seiten
![]() Home
Home
©
Copyright Text, Graphik und Photos: Bernhard Peter 2006
Impressum