
Bernhard
Peter
Schlüsselenzym
Protease: Die chemische Schere
Die HIV-Protease ist ein vom Virus codiertes Enzym, das langkettige, viral gebildete Polypeptide in kleinere Proteineinheiten zerschneidet, die dann zu infektiösen Virionen zusammengebaut werden. Bei der Herstellung neuer Viren wird zuerst die mittlerweile in den Zellkern eingebaute DNA abgelesen und eine lange Boten-RNA erstellt - wie bei der ganz normalen Transkription und Translation, nur daß der Virus die Kontrolle über den Zellstoffwechsel übernommen hat und alle zellulären Systeme in seinen Dienst gestellt hat. Anhand dieser werden an den Ribosomen der Zelle lange Polypeptidketten in Massen produziert, die quasi alle benötigten Teilstrukturen am Stück enthalten. Als Ganzes ist das nicht verwertbar, die lange Kette muß erst noch enzymatisch gespalten werden. Diese chemische Schere ist die Protease. Sie zerteilt das Primärprodukt in die einzelnen für den Zusammenbau neuer Viren benötigten Abschnitte: Matrixproteine, Hüllproteine, Enzyme, insgesamt 15 verschiedene.
Die HIV-Protease ist ein symmetrisches Homo-Dimer und besteht aus zwei identischen Untereinheiten mit einem gemeinsamen aktiven Zentrum. Das ist ein weiteres Beispiel für den effizienten Umgang mit Platz in den kleinen Virionen: So wenig Information wie möglich, so reduziert und minimalistisch wie möglich, möglichst wenig verschiedene Strukturen kodieren. Viren sind bewundernswert einfach und auf's Wesentliche beschränkt gebaut. Es ist ein kleines Enzym, jede Kette ist nur 99 Aminosäuren lang.

Abbildung: Dreidimensionale Struktur der Protease von HIV-1 (PDB-ID 1ODW) in sphärischer Darstellung. Das Enzym legt als Dimer vor, die beiden identischen Polypeptidketten sind in unterschiedlichen Farben dargestellt. Gemeinsam formen sie das aktive Zentrum (Hohlraum im unteren Bereich).
Wie man erkennt, sind die beiden Polypeptidketten im oberen Bereich der Abbildung relativ eng miteinander verzahnt und relativ fest durch nebenvalente Wechselwirkungen miteinander verbunden. Im unteren Bereich legen sich die beweglichen Teile wie Klammern um die zu schneidende Polypeptidkette und lassen nach dem Schnitt wieder los. Zur Spaltung liegt die Polypeptidkette in der Umarmung des Enzyms wie in einem Tunnel, in der obigen Abbildung von einer Mündung aus betrachtet.
Im Detail ist der Vorgang der Spaltung noch ein wenig differenzierter: Die HIV-Protease ist eine Aspartylprotease, wie auch z. B. das Pepsin oder das Renin. Das bedeutet, daß sich im aktiven Zentrum zwei Asparaginsäuren befinden, die für den Prozeß der Spaltung wichtig sind. Das aktive Zentrum befindet sich in einem zentralen Hohlraum, in die sich die zu spaltenden Polypeptidstränge hineinlegen, um dort Hydrolyse zu erfahren und dadurch gespalten zu werden. Lipophile Aminosäuren (Valin, Isoleucin und Leucin) ragen in den Hohlraum hinein und umgeben die Bindungshöhe. Anscheinend spaltet die Protease bevorzugt zwischen unpolaren Aminosäuren, z. B. zwischen Phenylalanin und Prolin, zwischen Phenylalanin und Leucin oder zwischen Phenylalanin und Threonin. Dabei dürfen diese nicht endständig sein und sie müssen mehrere unpolare Aminosäuren als Nachbarn aufweisen. Diese Substratspezifität kommt durch die vielen hydrophoben Aminosäuren im aktiven Zentrum der Protease zustande.
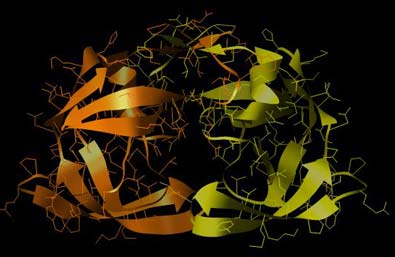
Abbildung: Dreidimensionale Struktur der Protease von HIV-1 (PDB-ID 1ODW). Das Enzym legt als Dimer vor, die beiden identischen Polypeptidketten sind in unterschiedlichen Farben dargestellt. Hervorgehoben ist hier die Sekundärstruktur (Helix, Faltblatt), die einzelnen Aminsäuren sind zusätzlich in Strichform abgebildet. Gemeinsam formen beide Ketten das aktive Zentrum (großer Hohlraum im unteren Bereich).
Literatur:
Hans-Dieter Höltje, Monika
Höltje, Struktur und Funktion von HIV-Protease-Hemmern,
Pharmazeutische Zeitung 27 (1997) 31-38
Research Collaboratory for Structural Bioinformatics Protein Data
Bank http://www.rcsb.org/pdb, PDB-ID: 1ODW: Native HIV-1
Protease, Kervinen, J., Thanki, N., Zdanov,
A., Tino, J., Barrish, J., Lin,
P.F., Colonno, R., Riccardi, K., Samanta,
H., Wlodawer, A. Structural Analysis of the Native and
Drug-Resistant HIV-1 Proteinases Complexed with an Aminodiol
Inhibitor Protein Pept.Lett. v3 pp.399 , 1996
David S. Goodsell, HIV-1 Protease:
http://www.rcsb.org/pdbstatic/education_discussion/molecule_of_the_month/download/HIV-1Protease.pdf,
http://www.rcsb.org/pdb/static.do?p=education_discussion/molecule_of_the_month/pdb6_1.html.
So
funktionieren HIV-Medikamente:
Ein Schlüsselenzym: Die Reverse
Transkriptase
So setzt man die Reverse Transkriptase
außer Gefecht: Antimetabolite
So setzt man die Reverse Transkriptase
außer Gefecht: nicht-nukleosidische Hemmstoffe
Schlüsselenzym Protease: Die chemische
Schere
Wir setzen die chemische Schere außer
Betrieb: Protease-Hemmstoffe
PI:
Protease-Inhibitoren
Indinavir, Nelfinavir, Saquinavir, Lopinavir, Amprenavir, Fosamprenavir, Ritonavir, Atazanavir, Tipranavir, DMP 450
Zurück zur Übersicht HIV / AIDS
Andere pharmazeutische Seiten
![]() Home
Home
©
Copyright Text, Graphik und Photos: Bernhard Peter 2006
Impressum