Bernhard
Peter
Wir
setzen die chemische Schere außer Betrieb: Protease-Inhibitoren
Diese Stoffe hemmen die Protease, das ist das Enzym, welches für den Schnitt der hergestellten Vorläufer-Proteine und für den Zusammenbau der Viren aus den synthetisierten neuen Bausteinen verantwortlich ist. Die Blockierung des Enzyms führt zu unreifen, nicht-infektiösen Viren. Protease-Inhibitoren verhindern deshalb neue Infektionszyklen. Dabei gibt es zwei Sorten Wirkstoffe, die eine Sorte sind selber z. T. Peptide, die andere nicht (nicht-peptidische Protease-Hemmer).
Die kompetitiven PI tun so, als ob sie die bevorzugte Schnittstelle wären und täuschen die Protease, die ihre Funktion dann nicht mehr ausführen kann, denn die falschen Moleküle haben an der „Möchtegern-Schnittstelle“, wo das Enzym spalten will, keine peptidische Bindung. Um effiziente Hemmstoffe zu haben, brauchen wir Moleküle mit unpolaren Gruppen mit hoher Affinität zur Innenauskleidung des Hohlraumes (aktives Zentrum), die aber an der entscheidenden Stelle keine spaltbare Bindung haben.
Eine besondere Rolle spielt bei der Spaltungh ein Wassermolekül im aktiven Zentrum, welches nur bei HIV-Proteasen und nicht bei anderen Aspartylproteasen gefunden wurde.
Die ersten Wirkstoffe lehnten sich noch sehr eng an die peptidartige Grundstruktur an. Vertreter sind Saquinavir, Indinavir und Ritonavir. Die Arzneistoffe sind alle relativ ähnlich, müssen sie auch sein, um die Eigenschaften des Kanals komplementär zu ergänzen und so optimal da rein zu passen. Mindestens zwei Sauerstoff-Funktionen im Zentrum des Kanals, die mit dem erwähnten Wassermolekül interagieren, dazwischen aber keine spaltbare Bindung, außen zu beiden Seiten des Tunnels kohlenstoffreiche und unpolare Teilstrukturen.

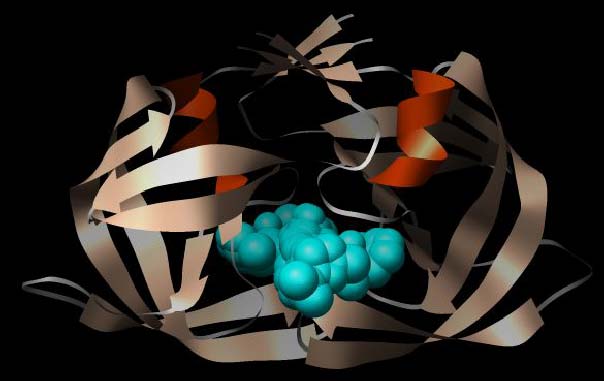
Abbildung: Dreidimensionale Struktur der Protease von HIV-1 (PDB-ID 1RL8) mit Ritonavir im aktiven Zentrum. Das Enzym legt als Dimer vor, gemeinsam formen die beiden identischen Ketten das aktive Zentrum (großer Hohlraum im unteren Bereich), das durch den Hemmstoff ausgefüllt und blockiert wird.
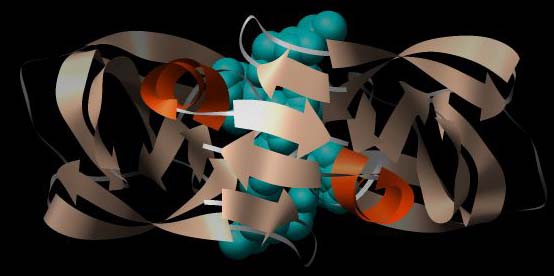
Abbildung: Dreidimensionale Struktur der Protease von HIV-1 (PDB-ID 1RL8) mit Ritonavir im aktiven Zentrum, im Gegensatz zur obigen Abbildung von oben.
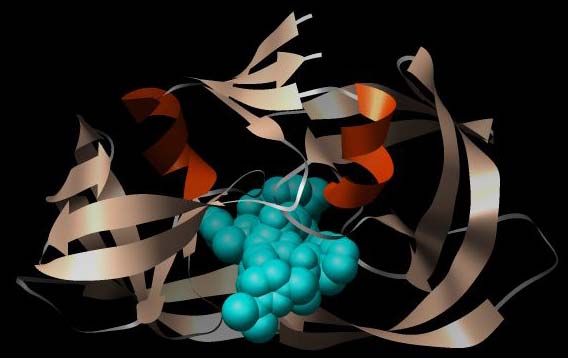
Abbildung: Dreidimensionale Struktur der Protease von HIV-1 (PDB-ID 1RL8) mit Ritonavir im aktiven Zentrum, im Gegensatz zur obigen Abbildung schräg von vorn.
Inzwischen geht die Entwicklung zu nicht-peptidischen Strukturen, weil man sich davon verbesserte Eigenschaften und vor allem eine höhere Selektivität und Affinität zum Enzym erhofft. Eine Modellstruktur ist das zyklische Harnstoff-Derivat DMP-450. Besonders interessant ist die Carbonylgruppe, die die Stelle des oben erwähnten Wassermoleküls einnimmt und für eine festere Bindung an das Enzym sorgt.
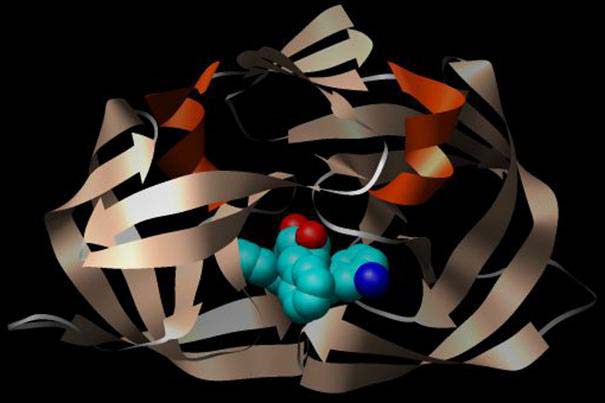
Abbildung: Dreidimensionale Struktur der Protease von HIV-1 (PDB-ID1MER) mit DMP 450 im aktiven Zentrum. Das Enzym legt als Dimer vor, gemeinsam formen die beiden identischen Ketten das aktive Zentrum (großer Hohlraum im unteren Bereich), das durch den Hemmstoff ausgefüllt und blockiert wird.
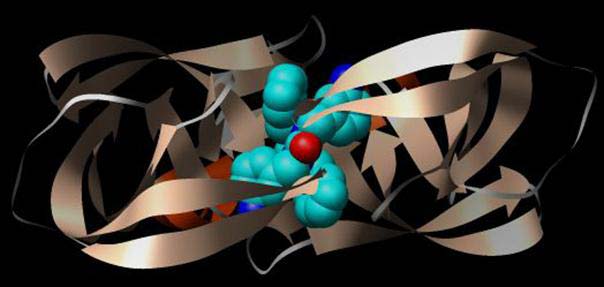
Abbildung: Dreidimensionale Struktur der Protease von HIV-1 (PDB-ID1MER) mit DMP 450 im aktiven Zentrum, im Gegensatz zur obigen Abbildung von unten.
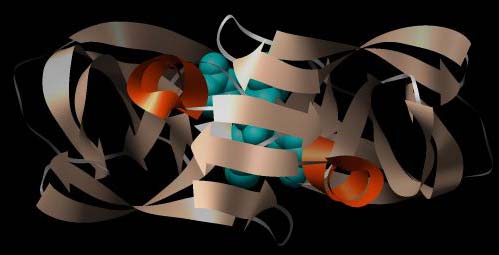
Abbildung: Dreidimensionale Struktur der Protease von HIV-1 (PDB-ID1MER) mit DMP 450 im aktiven Zentrum, im Gegensatz zur obigen Abbildung von oben.
Der große Vorteil der Protease-Hemmer gegenüber den Antimetaboliten (NRTI, NtRTI und NNRTI) liegt im späteren Angriffspunkt. Wenn die Virus-RNA erst einmal in DNA umgeschrieben und in das Genom insertiert ist, ist bei den Antimetaboliten der Zug abgefahren: Man kann nur die Infektion neuer Zellen verhindern, muß aber tatenlos zusehen, wie bereits infizierte Zellen weiter lustig neue Viren produzieren. Protease-Hemmer dagegen inhibieren auch die Vermehrung von Viren aus Zellen, in deren Genom die Virus-Information bereits insertiert ist. Ein weiterer entscheidender Vorteil der Protease-Hemmer gegenüber den Antimetaboliten ist die höhere Selektivität und die geringere mitochondriale Toxizität. Dem stehen aber andere Nebenwirkungen insbesondere im Lipidstoffwechsel entgegen (siehe einzelne Verbindungen).
Das Problem der Resistenzbildung gilt leider auch für die Protease-Inhibitoren. Ihren Stellenwert haben die PIs heute vor allem in HAART-Kombinationstherapien.
Literatur:
Hans-Dieter Höltje, Monika
Höltje, Struktur und Funktion von HIV-Protease-Hemmern,
Pharmazeutische Zeitung 27 (1997) 31-38
Research Collaboratory for Structural Bioinformatics Protein Data
Bank http://www.rcsb.org/pdb, PDB-ID: 1 MER, HIV-1 MUTANT (I84V)
PROTEASE COMPLEXED WITH DMP450, Ala, P.J., Huston,
E.E., Klabe, R.M., McCabe, D.D., Duke,
J.L., Rizzo, C.J., Korant, B.D., DeLoskey,
R.J., Lam, P.Y., Hodge, C.N., Chang, C.H.
Molecular basis of HIV-1 protease drug resistance: structural
analysis of mutant proteases complexed with cyclic urea
inhibitors. Biochemistry v36 pp.1573-1580 , 1997
Research Collaboratory for Structural Bioinformatics Protein Data
Bank http://www.rcsb.org/pdb, PDB-ID: 1RL8, Crystal structure of
the complex of resistant strain of hiv-1 protease(v82a mutant)
with ritonavir Rezacova, P., Brynda,
J., Sedlacek, J., Konvalinka, J., Fabry,
M., Horejsi, M. Crystal structure of the complex of
resistant strain of hiv-1 protease(v82a mutant) with ritonavir,
in Publikation, 2005
So
funktionieren HIV-Medikamente:
Ein Schlüsselenzym: Die Reverse
Transkriptase
So setzt man die Reverse Transkriptase
außer Gefecht: Antimetabolite
So setzt man die Reverse Transkriptase
außer Gefecht: nicht-nukleosidische Hemmstoffe
Schlüsselenzym Protease: Die chemische
Schere
Wir setzen die chemische Schere außer
Betrieb: Protease-Hemmstoffe
PI:
Protease-Inhibitoren
Indinavir, Nelfinavir, Saquinavir, Lopinavir, Amprenavir, Fosamprenavir, Ritonavir, Atazanavir, Tipranavir, DMP 450
Zurück zur Übersicht HIV / AIDS
Andere pharmazeutische Seiten
![]() Home
Home
©
Copyright Text, Graphik und Photos: Bernhard Peter 2006
Impressum